Dihydrolipoamid S- Succinyltransferase
| Dihydrolipoamid S- Succinyltransferase | |||||||||||||||||
 Struktur einer E. coli- Dihydrolipoamid- S-Succinyltransferase ( PDB 1C4T ) | |||||||||||||||||
| Haupteigenschaften | |||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Symbol | DLST | ||||||||||||||||
| EG-Nr. | 2.3.1.61 | ||||||||||||||||
| Homo sapiens | |||||||||||||||||
| Ort | 14 q 24.3 | ||||||||||||||||
| Molekulargewicht | 48 755 Da | ||||||||||||||||
| Anzahl der Rückstände | 453 Aminosäuren | ||||||||||||||||
| Links über GeneCards und HUGO zugänglich . | |||||||||||||||||
| |||||||||||||||||
Die Dihydrolipoamid- S- Succinyltransférase ist eine Acyltransferase, die Teil des α-Ketoglutarat-Dehydrogenase-Komplexes ist , wobei es sich um den Enzym- E2- Komplex handelt, der die Umwandlung von α-Ketoglutarat in Succinyl-CoA und CO 2 durchführt ::
|
|
+ CoA-SH + NAD + → NADH + H + + CO 2 + |
|
| α-Ketoglutarat | Succinyl-CoA |
Genauer gesagt katalysiert das Enzym E2 die Übertragung der Succinylgruppe auf Coenzym A von dem Lipoamid, an das es im vorherigen Schritt fixiert wurde, katalysiert durch α-Ketoglutarat-Dehydrogenase .
Der Mechanismus dieser Reaktion, an der nacheinander die Enzyme E1, E2 und E3 mit ihren Cofaktoren beteiligt sind , ist recht komplex und kann durch das folgende vereinfachte Diagramm zusammengefasst werden:
Dihydrolipoamid S- Succinyltransferase
Dihydrolipoamid- S- Succinyltransferase ist eine Acyltransferase, an der nacheinander drei Cofaktoren beteiligt sind : Thiaminpyrophosphat (TPP, gebunden an Alpha-Ketoglutarat-Dehydrogenase ), Dihydrolipoamid und Coenzym A (CoA-SH), wobei letzteres in Acetyl-Coenzym A (Acetyl) umgewandelt wird.

|

|

|
||
| Thiaminpyrophosphat (TPP) | Dihydrolipoamid | Coenzym A (CoA-SH) |
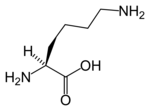
Der wahre Cofaktor ist eigentlich Liponsäure mehr als (dihydro) Lipoamid, aber diese Carbonsäure ist , kovalent gebunden an das E2 - Protein mittels einer Amid - Bindung an einem Lysin - Rest , in dem äquivalent von ‚einem Rest von Lipoamid führt.
DihydrolipoamidS- Succinyltransferase
| EG-Nr. | EG |
|---|---|
| CAS-Nummer |
| IUBMB | IUBMB-Eintrag |
|---|---|
| IntEnz | IntEnz-Ansicht |
| BRENDA | BRENDA Eingang |
| KEGG | KEGG-Eingang |
| MetaCyc | Stoffwechselweg |
| PRIAM | Profil |
| PDB | Strukturen |
| GEHEN | AmiGO / EGO |

|
|
|
| Liponsäure | L- Lysin |
Anmerkungen und Referenzen
- (in) James E. Knapp Donald Carroll, Janet E. Lawson, Stephen R. Ernst, Lester J. Reed und Marvin L. Hackert , " Expression, Reinigung und Strukturanalyse der trimeren Form der katalytischen Domäne der Escherichia coli Dihydrolipoamid-Succinyltransferase ” , Protein Science , vol. 9, n o 1, Januar 2000, p. 37-48 ( PMID 10739245 , PMCID 2144448 , DOI 10.1110 / ps.9.1.37 , online lesen )
- Die hier angegebenen Werte für die Masse und die Anzahl der Reste sind diejenigen des Proteinvorläufers, die aus der Translation des Gens vor posttranslationalen Modifikationen resultieren , und können sich erheblich von den entsprechenden Werten für die funktionelles Protein .